BIOLOGÍA GRADO 9º
REPLICACION DEL ADN
Una vez que se comprobó que el ADN era el material hereditario
y se descifró su estructura, lo que quedaba era determinar como el ADN copiaba
su información y como la misma se expresaba en el fenotipo. Matthew Meselson y
Franklin W. Stahl diseñaron el experimento para determinar el método de
la replicación del
ADN. Tres modelos de replicación era plausibles.
1. Replicación conservativa durante la
cual se produciría un ADN completamente nuevo durante la replicación.

2. En la replicación semiconservativa se
originan dos moléculas de ADN, cada una de ellas compuesta de una hebra de el
ADN original y de una hebra complementaria nueva. En otras palabras el ADN se
forma de una hebra vieja y otra nueva. Es decir
que las hebras existentes sirven de molde complementario a las
nuevas.




3. La replicación dispersiva implicaría
la ruptura de las hebras de origen durante la replicación que, de alguna manera
se reordenarían en una molécula con una mezcla de fragmentos nuevos y viejos en
cada hebra de ADN.
El experimento de Meselson-Stahl
consiste en cultivar la bacteria Escherichia coli en un medio que
contenga nitrógeno pesado (15Nitrógeno que es mas pesado que el isótopo mas
común: el 14Nitrógeno ). La primera generación de bacterias se hizo crecer
en un medio que únicamente contenía 15Nitrógeno como fuente de N. La
bacteria se transfirió luego a un medio con 14N. Watson y Crick habían
pronosticado que la replicación del ADN era semiconservativa, de ser así el ADN
extraído de las bacterias luego de cultivarlas por una generación en 14N
tendría un peso intermedio entre el ADN extraído del medio con 15N y el
del extraído de medio con 14N y así fue.
.
Los detalles del experimento que
incluye un proceso de ultracentrifugación en cloruro de Cesio (CeCl2) puede
encontrarse en el Curtis.
La replicación del ADN, que
ocurre una sola vez en cada generación celular, necesita de muchos
"ladrillos", enzimas, y una gran cantidad de energía en forma de ATP
(recuerde que luego de la fase S del ciclo celular las
células pasan a una fase G a fin de, entre otras cosas, recuperar
energía para la siguiente fase de la división celular). La replicación del ADN
en el ser humano a una velocidad de 50 nucleótidos por segundo, en procariotas
a 500/segundo. Los nucleótidos tienen que se armados y estar disponibles en el
núcleo conjuntamente con la energía para unirlos.
La iniciación de la replicación
siempre acontece en un cierto grupo de nucleótidos, el origen de la
replicación, requiere entre otras de las enzimas helicasas para
romper los puentes hidrógeno y las topoisomerasas para aliviar la
tensión y de las proteínas de unión a cadena simple para mantener
separadas las cadenas abiertas.

Una vez que se abre la molécula,
se forma una área conocida como "burbuja de replicación" en ella se
encuentran las "horquillas de replicación" . Por acción de la la ADN
polimerasa los nuevos nucleótidos entran en la horquilla y se enlazan con el
nucleótido correspondiente de la cadena de origen (A con T, C con G). Los
procariotas abren una sola burbuja de replicación, mientras que los eucariotas
múltiples. El ADN se replica en toda su longitud por confluencia de las
"burbujas".
Dado que las cadenas del ADN son
antiparalelas, y que la replicación procede solo en la dirección 5' to 3'
en ambas cadenas, numerosos experimentos mostraron que, una cadena formará una
copia continua, mientras que en la otra se formarán una serie de fragmentos
cortos conocidos como fragmentos de Okazaki . La cadena que se sintetiza de
manera continua se conoce como cadena adelantada y, la que se
sintetiza en fragmentos, cadena atrasada.
Para que trabaje la ADN
polimerasa es necesario la presencia, en el inicio de cada nuevo fragmento, de
pequeñas unidades de ARN conocidas como cebadores, a posteriori, cuando la polimerasa toca
el extremo 5' de un cebador, se activan otras enzimas, que remueven los
fragmentos de ARN, colocan nucleótidos de ADN en su lugar y, una ADN ligasa los
une a la cadena en crecimiento.
Tomado de: http://www.biologia.edu.ar/adn/adntema1.htm
VÍDEO COMPLEMENTARIO
VÍDEO COMPLEMENTARIO
TRANSCRIPCIÓN DEL ADN
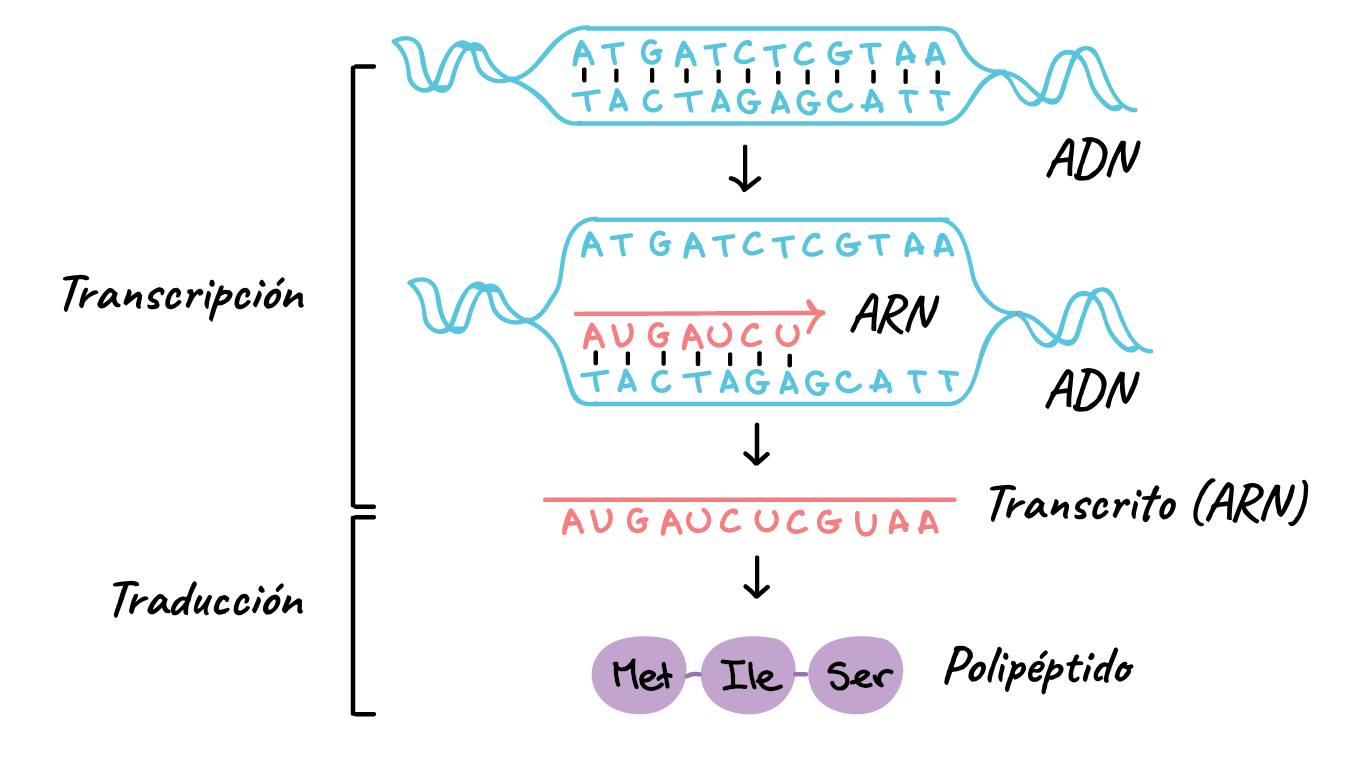
La transcripción es el primer
paso de la expresión génica, el proceso por el cual la información de un gen se utiliza para generar un producto funcional, como una
proteína. El objetivo de la transcripción es producir una copia de ARN de la
secuencia de ADN de un gen. En el caso de los genes codificantes, la copia de
ARN, o transcrito, contiene la información necesaria para
generar un polipéptido (una proteína o la subunidad de una proteína). Los
transcritos eucariontes necesitan someterse a algunos pasos de procesamiento
antes de traducirse en proteínas.
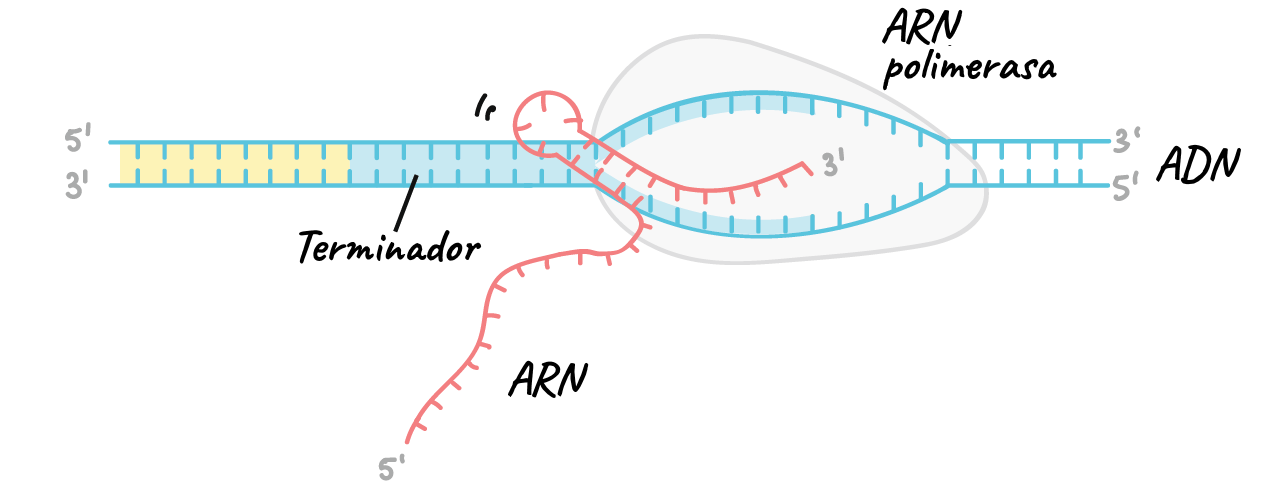

- La ARN polimerasa
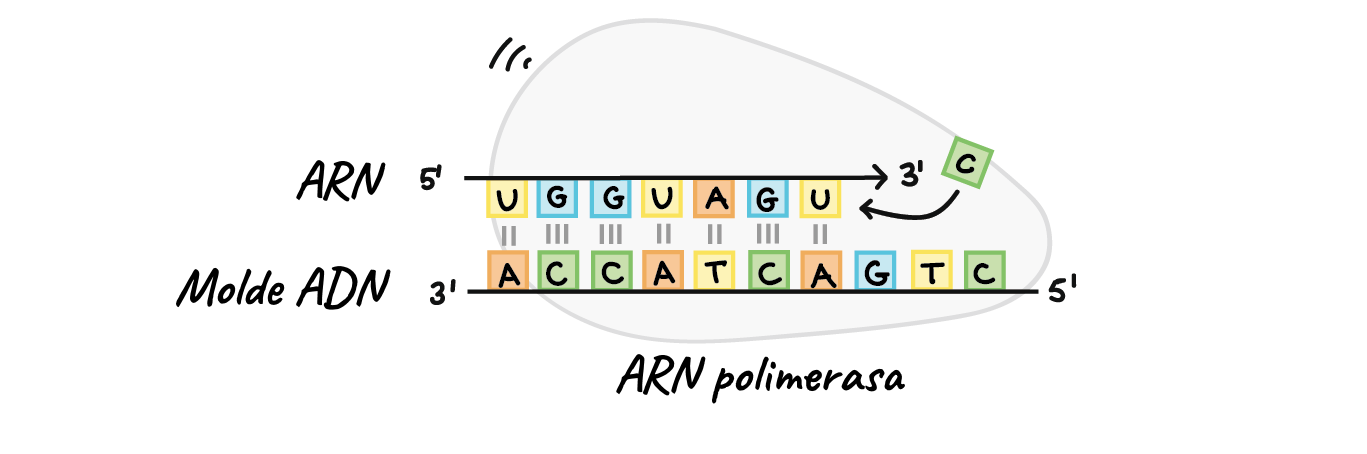
La principal enzima que participa en la transcripción es
la ARN polimerasa, la cual utiliza un molde de ADN de cadena sencilla para
sintetizar una cadena complementaria de ARN. Específicamente, la ARN polimerasa
produce una cadena de ARN en dirección de 5' a 3', al agregar cada nuevo
nucleótido al extremo 3' de la cadena.

Las etapas de la transcripción
La transcripción de un gen ocurre
en tres etapas: iniciación, elongación y terminación. Aquí veremos brevemente
cómo ocurren estas etapas en bacterias. Puedes aprender más sobre los detalles
de cada etapa (y sobre las diferencias que hay respecto a la transcripción
eucarionte) en el artículo sobre etapas de la transcripción.
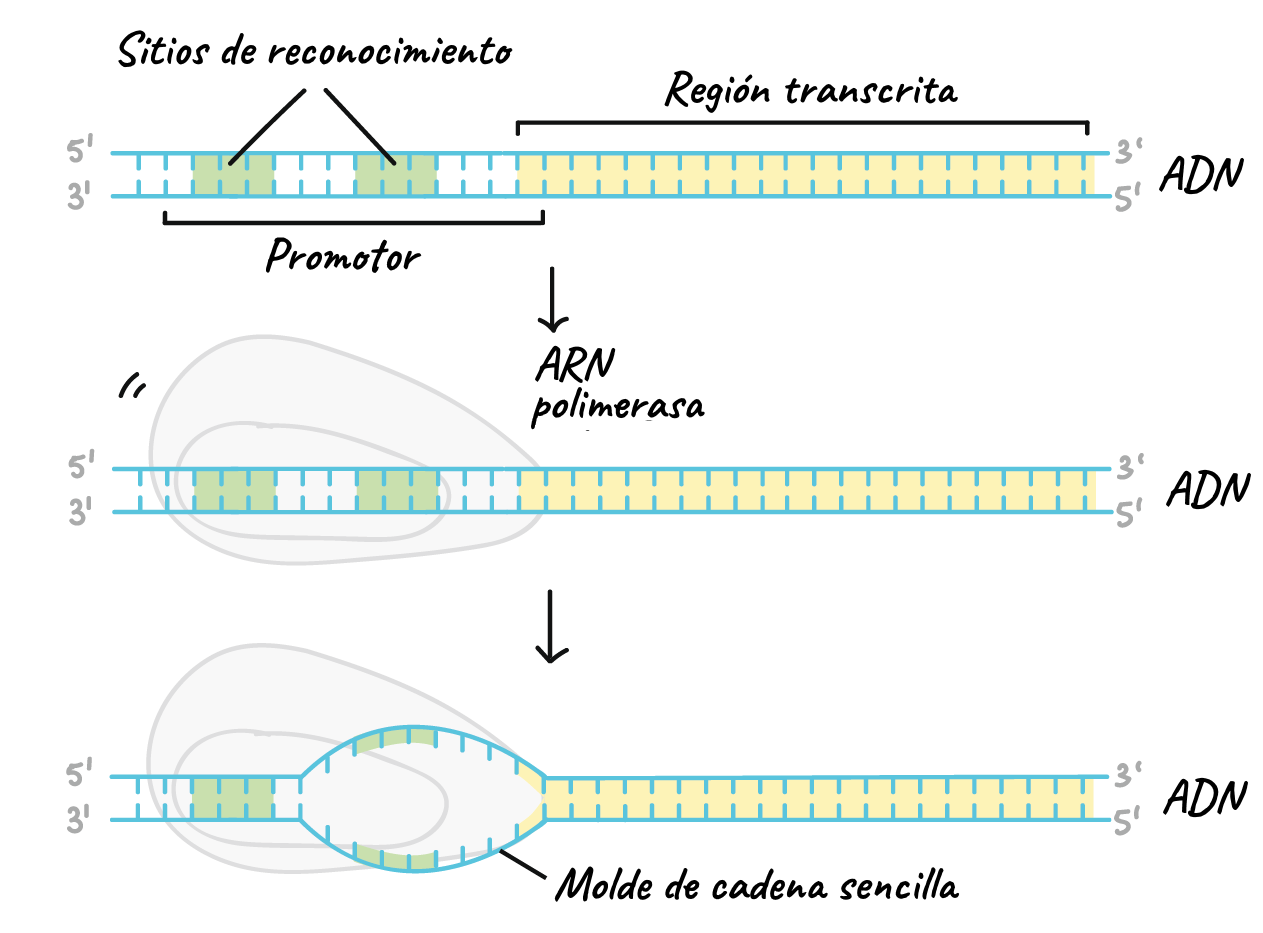
- Iniciación. La ARN polimerasa se une a una secuencia de ADN llamada promotor, que se encuentra al inicio de un gen. Cada gen (o grupo de genes co-transcritos en bacterias) tiene su propio promotor. Una vez unida, la ARN polimerasa separa las cadenas de ADN para proporcionar el molde de cadena sencilla necesario para la transcripción.

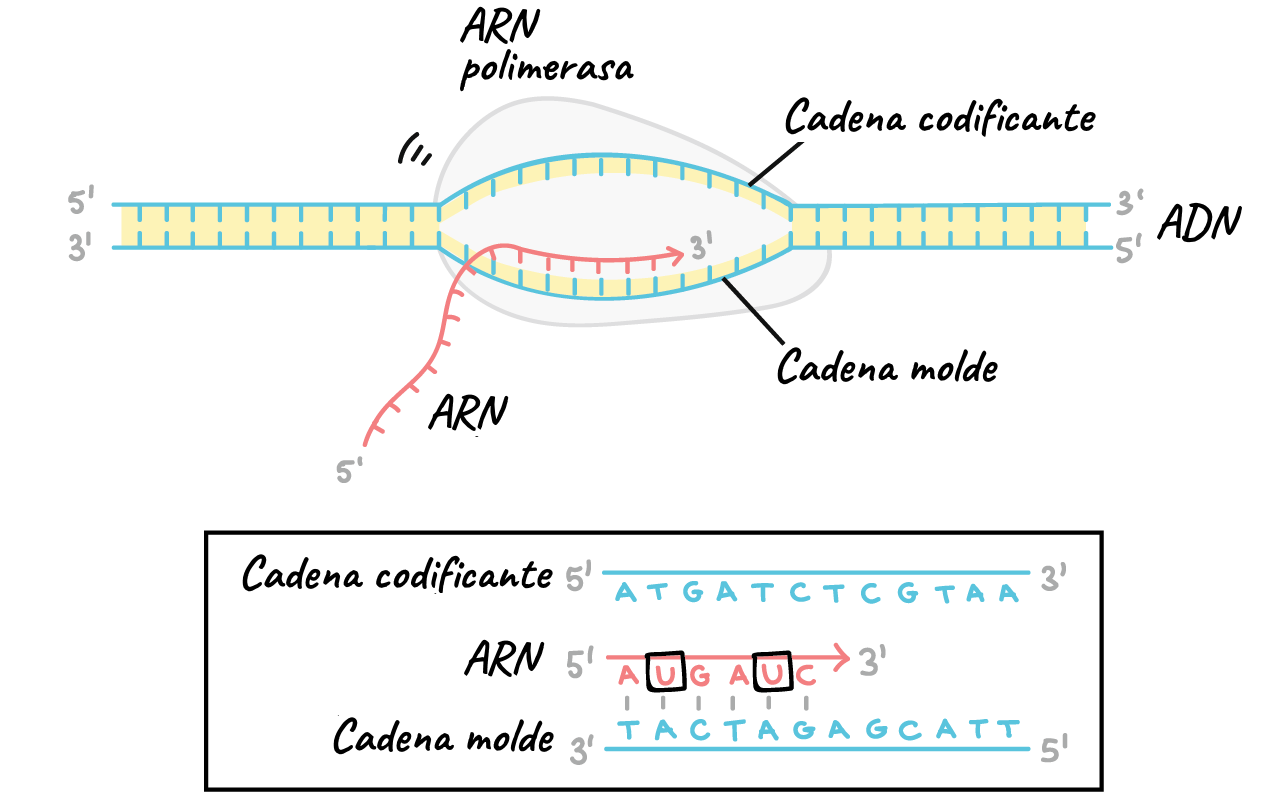
- Elongación. Una cadena de ADN, la cadena molde, actúa como plantilla para la ARN polimerasa. Al "leer" este molde, una base a la vez, la polimerasa produce una molécula de ARN a partir de nucleótidos complementarios y forma una cadena que crece de 5' a 3'. El transcrito de ARN tiene la misma información que la cadena de ADN contraria a la molde (codificante) en el gen, pero contiene la base uracilo (U) en lugar de timina (T).

- Terminación. Las secuencias llamadas terminadores indican que se ha completado el transcrito de ARN. Una vez transcritas, estas secuencias provocan que el transcrito sea liberado de la ARN polimerasa. A continuación se ejemplifica un mecanismo de terminación en el que ocurre la formación de un tallo-asa en el ARN

- MODIFICACIONES AL ARN EUCARIONTE
En bacterias, los transcritos de
ARN pueden actuar como ARN mensajeros (ARNm) inmediatamente. En
eucariontes, el transcrito de un gen codificante se llama pre-ARNm y
debe experimentar un procesamiento adicional antes de que pueda dirigir la
traducción.
Los pre-ARNm eucariontes deben
tener sus extremos modificados por la adición de un cap 5' (al
inicio) y una cola de poli-A 3' (al final).
Muchos pre-ARNm eucariontes
sufren empalme. En este proceso, partes del pre-ARNm (llamadas intrones)
se cortan y se eliminan, y las piezas restantes (llamadas exones) se
vuelven a unir.
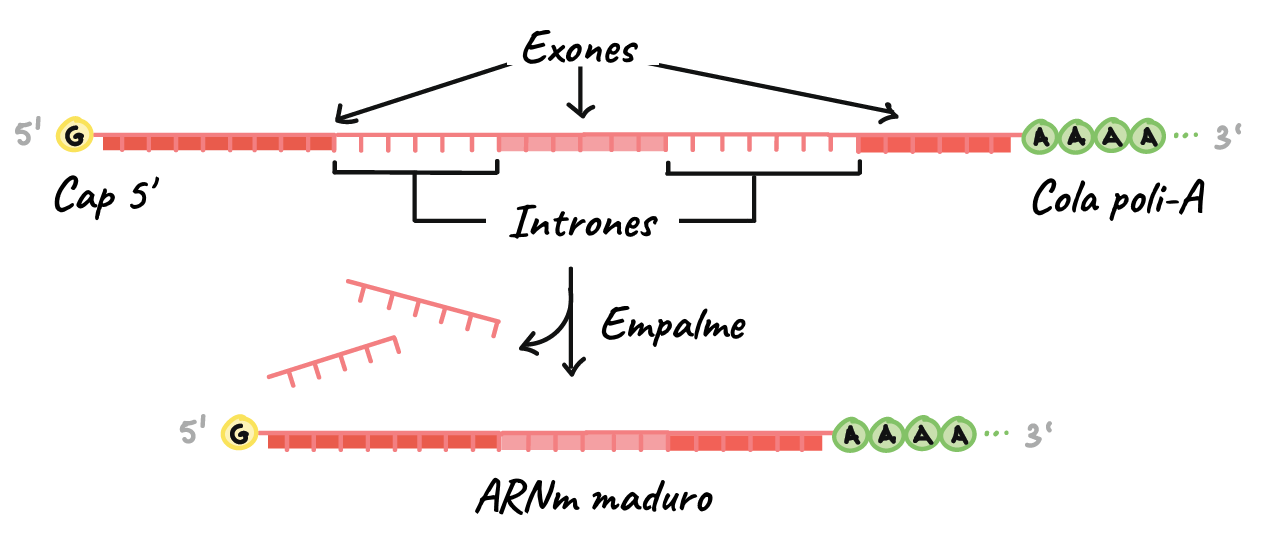

Las modificaciones en los
extremos aumentan la estabilidad del ARNm, mientras que el empalme otorga al
ARNm su secuencia correcta (si no se eliminan los intrones, se traducirán junto
con los exones y producirán un polipéptido "sin sentido").
Tomado de: https://es.khanacademy.org/science/biology/gene-expression-central-dogma/transcription-of-dna-into-rna/a/overview-of-transcription
VÍDEO COMPLEMENTARIO
TRADUCCIÓN
Tomado de: https://es.khanacademy.org/science/biology/gene-expression-central-dogma/transcription-of-dna-into-rna/a/overview-of-transcription
VÍDEO COMPLEMENTARIO
TRADUCCIÓN
¿Cómo se
"lee" un ARNm para formar un polipéptido? Dos tipos de molécula con
papeles clave en la traducción son los ARNt y los ribosomas.
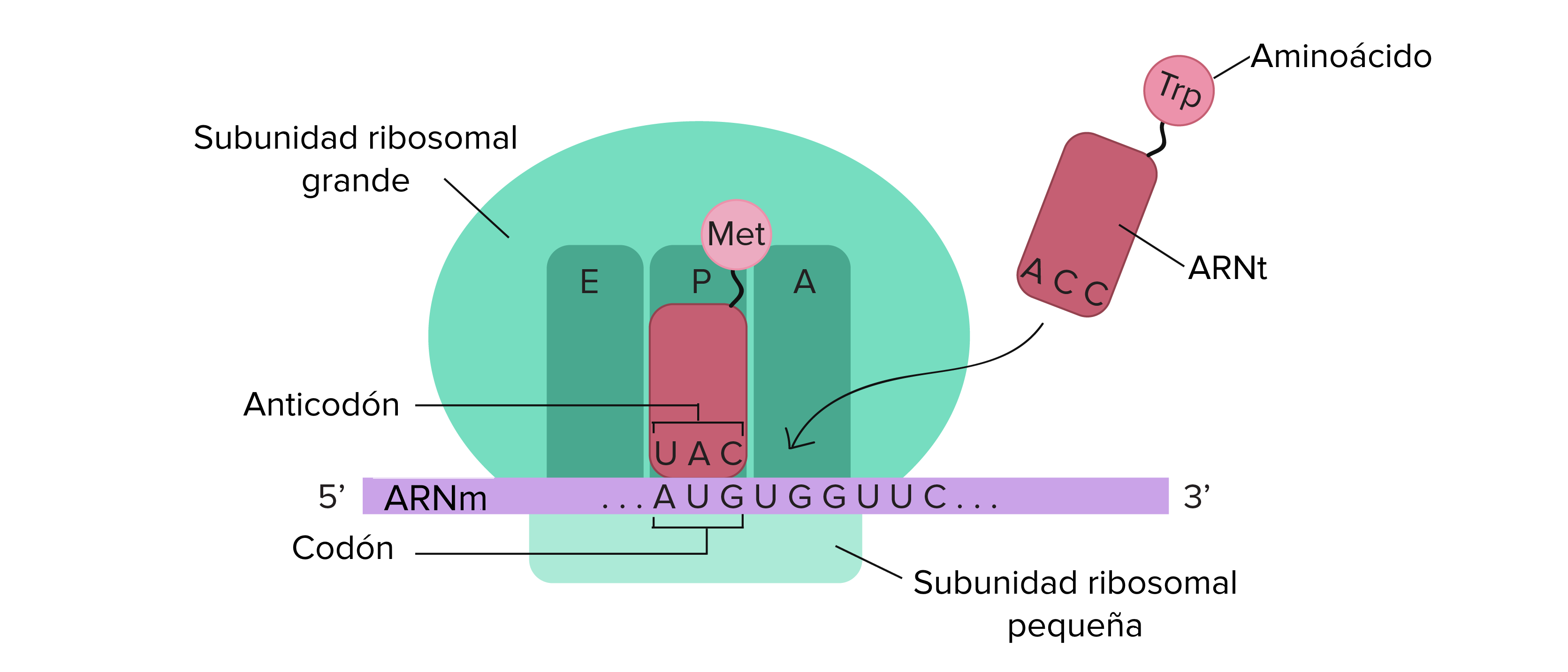
- ARNs de
transferencia (ARNt)
Los ARN
de transferencia o ARNt, son "puentes" moleculares que
conectan los codones del ARNm con los aminoácidos para los que codifican. Un
extremo de cada ARNt tiene una secuencia de tres nucleótidos
llamada anticodón, que se puede unir a un codón específico del ARNm. El
otro extremo de ARNt lleva el aminoácido que especifica el codón.
Hay
muchos tipos de ARNt. Cada tipo lee uno o unos pocos codones y lleva el
aminoácido correcto que corresponde a esos codones.

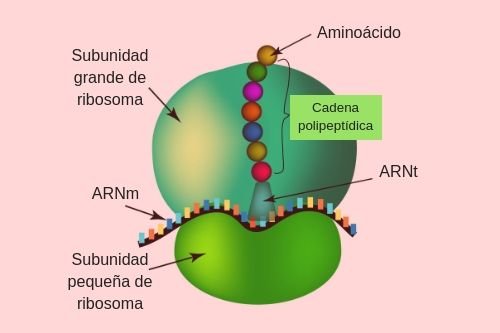
- Ribosomas
Los ribosomas son
las estructuras donde se construyen los polipéptidos (proteínas). Se componen
de proteínas y ARN (ARN ribosomal o ARNr). Cada ribosoma tiene dos
subunidades, una grande y una pequeña, que se reúnen alrededor de un ARNm, algo
parecido a las dos mitades de un pan para hamburguesa que se reúnen alrededor
de la torta de carne.
El
ribosoma proporciona un conjunto de espacios útiles o huecos donde los ARNt
pueden encontrar sus codones correspondientes en la plantilla del ARNm y
entregar sus aminoácidos. Estos huecos se llaman los sitios A, P y E. Pero
además el ribosoma actúa como una enzima que cataliza la reacción química que
une los aminoácidos para formar una cadena.

ETAPAS DE
LA TRADUCCIÓN
Tus
células están fabricando proteínas cada segundo, y cada una de ellas debe
contener el conjunto correcto de aminoácidos unidos justo en el orden debido.
Esto puede sonar como una tarea difícil, pero por suerte, tus células (junto
con las de los demás animales, plantes y bacterias) están capacitados para
ella.
Para ver
cómo las células hacen las proteínas, vamos a dividir la traducción en tres
etapas: iniciación (el comienzo), elongación (el agregar a la cadena proteica)
y terminación (la finalización).
- El comienzo: la iniciación
- La extensión de la cadena: elongación
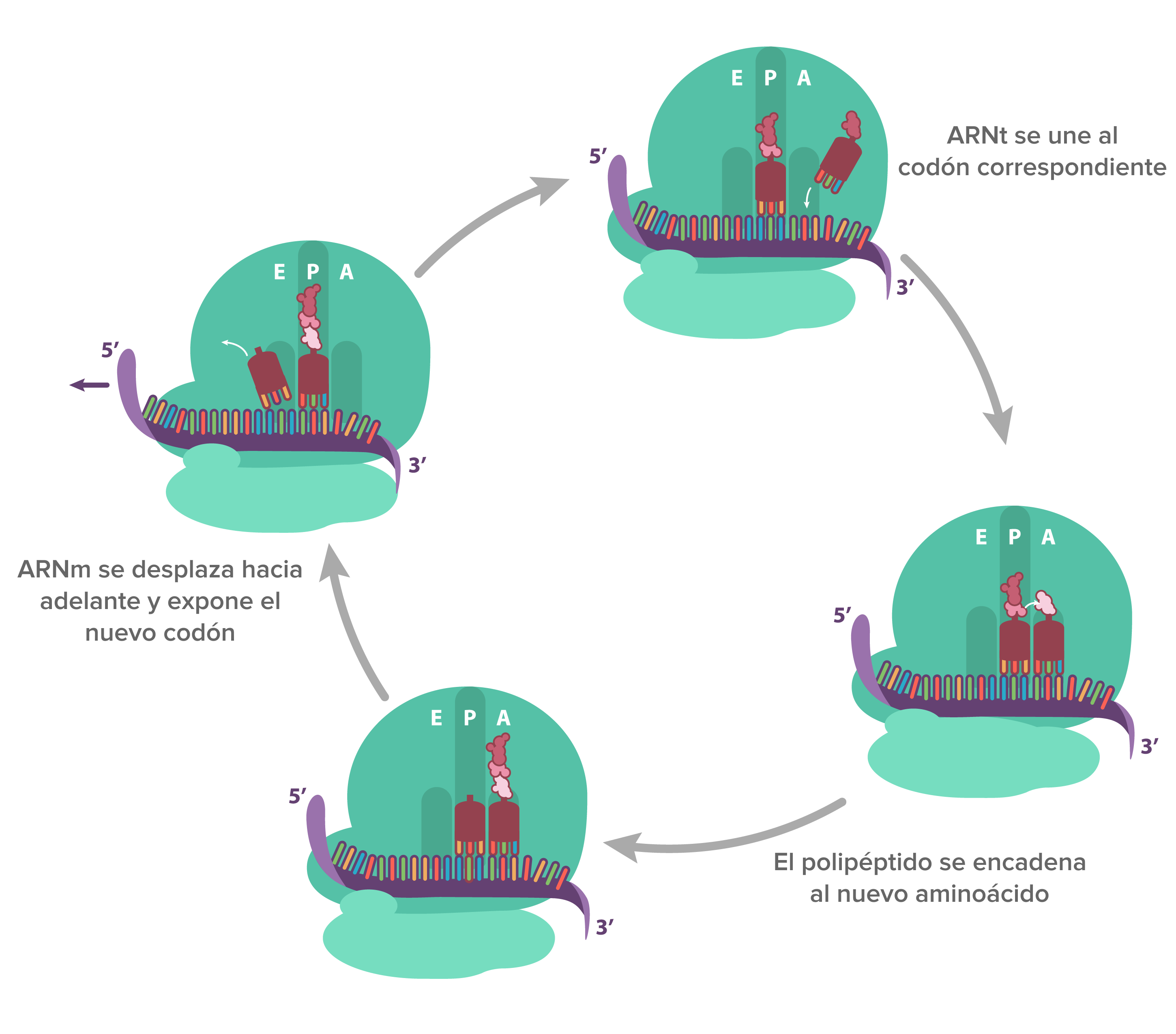
Cada vez
que un codón nuevo está expuesto:
- Un ARNt correspondiente se une al codón
- La cadena de aminoácidos existente (polipéptido) se une al aminoácido del ARNt mediante una reacción química.
- El ARNm se desplaza un codón sobre el ribosoma, lo que exponde un nuevo codón para que se lea.

Durante
la elongación, los ARNt pasan por los sitios A, P, y E como se muestra arriba.
Este proceso se repite muchas veces conforme se leen los nuevos codones y se
agregan los nuevos aminoácidos a la cadena.
- Finalizando el
proceso: terminación
La terminación es
la etapa donde la cadena polipeptídica completa es liberada. Comienza cuando un
codón de terminación (UAG, UAA o UGA) entra al ribosoma, lo que dispara una
serie de eventos que separa la cadena de su ARNt y le permite flotar hacia
afuera.
Después
de la terminación, es posible que el polipéptido todavía necesite tomar la
forma tridimensional correcta, se someta a procesamiento (tal como el retiro de
aminoácidos), sea enviado a la parte correcta en la célula, o se combine
con otros polipéptidos antes de que pueda hacer su trabajo como una proteína
funcional.
VÍDEO
COMPLEMENTARIO

No hay comentarios.:
Publicar un comentario